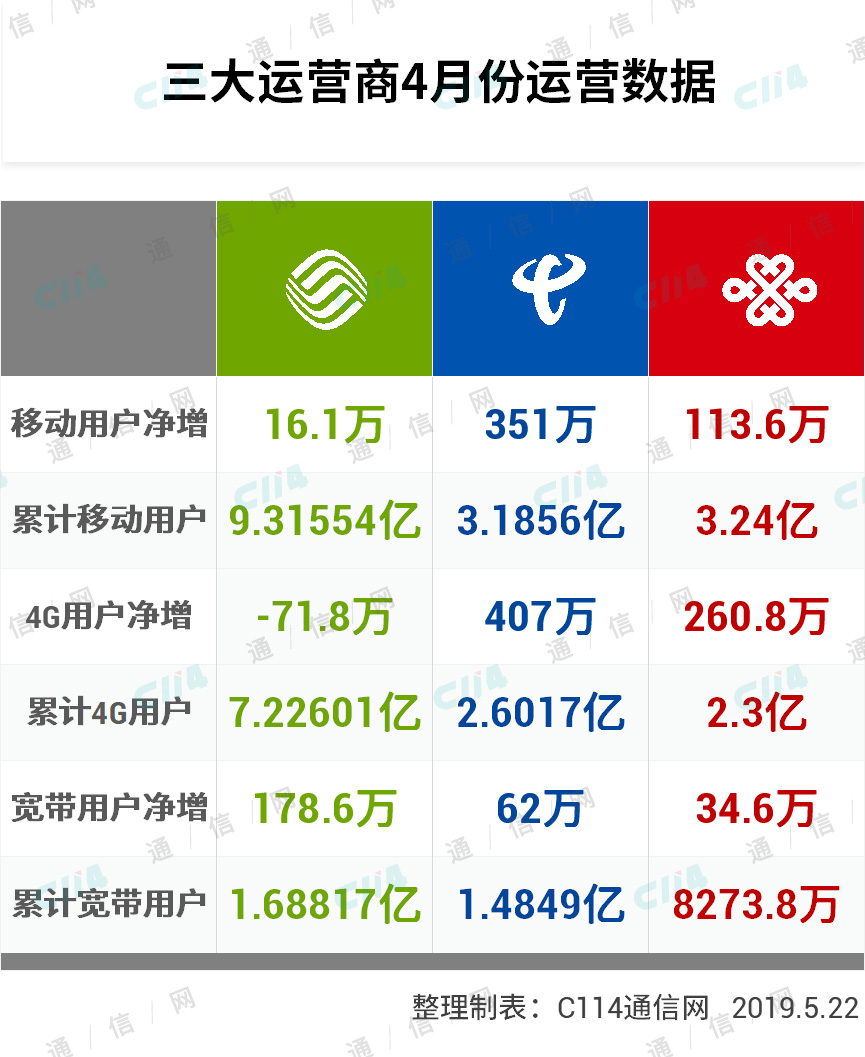
英国“深度思维”公司今年8月曾宣布,其开发的人工智能(AI)程序“阿尔法折叠”已预测出约100万个物种的超过2亿种蛋白质的结构,涵盖科学界已编录的几乎每一种。现在,元宇宙平台公司(Meta)研究人员利用人工智能ESMFold预测了来自细菌、病毒和其他尚未被表征微生物的6亿多种蛋白质的结构。相关研究已经提交生物预印本网站biorxiv.org。

在最新研究中,Meta AI蛋白质团队使用“大型语言模型”预测了这些蛋白质的结构。要构建能通过几个字母或单词预测文本的工具,离不开大型语言模型的基础。语言模型通常需要大量文本进行训练,为将其应用于蛋白质,研究团队用已知的蛋白质序列训练它们,这些已知蛋白质可由20个不同氨基酸组成的链来表达,每个氨基酸由一个字母表示。然后,ESMFold学会了用模糊的氨基酸比例“自动完成”蛋白质。
团队负责人亚历山大·里维斯表示,这些训练让ESMFold对包含蛋白质形状信息的蛋白质序列有了直观了解。而且,与“阿尔法折叠”一样,这一网络能将这些了解到的信息与已知蛋白质结构和序列之间关系的信息结合,生成预测结构。
随后,他们在一个数据库上运用这一模型,该数据库由来自土壤、海水、人类肠道、皮肤和其他微生物栖息地的“宏基因组”DNA组成,其中绝大多数DNA条目编码潜在蛋白质,来自从未被培养过也不为人所知的生物体。总体而言,ESMFold预测了超6.17亿种蛋白质的结构,且只花了两周时间。而“阿尔法折叠”预测一种结构可能需要几分钟。
团队指出,ESMFold虽然不像“阿尔法折叠”那么准确,但在预测结构方面的速度要快60倍,这意味着他们可将结构预测扩展到更大的数据库。
里维斯表示:“这些来自土壤、海水和人体的微生物分子是我们了解最少的结构,最新研究有助于我们更进一步洞悉生物学。”(科技日报记者 刘霞)